- Пиросеквенирование
-
Пиросеквенирование это метод секвенирования ДНК (определение последовательности нуклеотидов в молекуле ДНК), основанный на принципе «секвенирование путем синтеза». При включении нуклеотида происходит детекция высвобождающихся пирофосфатов.[1] Технология была разработана Полом Ниреном (Pål Nyrén) и его студентом Мустафой Ронаги, в Королевском Техническом Институте (Стокгольм) в 1996 году.[2][3][4]
Содержание
Процедура
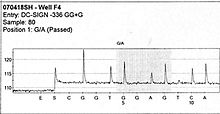
"Секвенирование путем синтеза" заключается в том, что для секвенирования одноцепочечной ДНК ферментативно синтезируют комплементарную цепочку. Метод пиросеквенирования основан на детекции активности фермента ДНК-полимеразы с другим хемилюминесцентным ферментом. Метод позволяет секвенировать одну цепочку нуклеотидов ДНК путем синтеза комплементарной цепочки, при этом регистрируется присоединение каждого нуклеотида. Матрица ДНК иммобилизована, растворы нуклеотидов A, C, G и T добавляются и отмываются последовательно после реакции. Свет образуется в тот момент, когда раствор нуклеотидов соответствует первому неспаренному основанию матрицы. Последовательность растворов, которые дают хемилюминесцентный сигнал, позволяет определить последовательность матрицы.
Матрица одноцепочечной ДНК гибридизуется с праймером и инкубируется с ферментами ДНК-полимеразой, АТФ-сульфурилазой, люциферазой и апиразой, а также с субстратами аденозин-5´-фосфосульфатами (APS) и люциферином.
- Добавление одного из четырех дезоксинуклеозидтрифосфатов (dNTP)(в случае dATP добавляют dATPαS, который не является субстратом для люциферазы) инициирует следующий этап. ДНК-полимераза включает правильный комплементарный дезокси-нуклеотид в цепочку. При этом стехиометрически высвобождается пирофосфат (PPi).
- Фермент АТФ-сульфурилаза количественно превращает PPi в аденозинтрифосфат (АТФ) в присутствии аденозин-5´-фосфосульфата. АТФ выступает "топливом" для фермента люциферазы, которая превращает люциферин в оксилюциферин, при этом высвобождается видимый свет, интенсивность которого пропорциональна количеству образовавшегося АТФ, Свет образуется в реакции, катализируемой люциферазой, регистрируется камерой и далее анализируется специальной компьютерной программой.
- Невключенные нуклеотиды и АТФ подвергаются деградации ферментом апиразой, и реакция начинается с новым нуклеотидом.
В настоящий момент, существуют некоторые ограничения в применения данного способа секвенирования. Лимитирующим фактором является длина последовательности нуклеотидов, которая составляет около 300-500 нуклеотидов, что короче, чем 800-1000 нуклеотидов, достижимые методом обрыва цепи (например, метод Сэнгера). Такие ограничения могут затруднять секвенирование геномов, в частности, богатых повторенными последовательностями нуклеотидов. К 2007 году, пиросеквенирование обычно использовали для повторного секвенирования или секвенирования геномов, для которых известна последовательность нуклеотидов родственного вида.
Коммерциализация
Компания "Pyrosequencing AB", которая располагается в Упсале, Швеция, коммерциализовала технологию и реагенты для секвенирования коротких участков ДНК. В 2003 году "Pyrosequencing AB" переименована в "Biotage". В 2008 году Qiagen приобрел Biotage.[5] Далее технология пиросеквенирования была лицензирована 454 Life Sciences. 454 разработала технологию пиросеквенирования, которая взяла начало от высокоэффективного секвенирования (High-throughput sequencing). Наиболее значимы приложения для секвенирования геномов и метагеномики. "GS FLX", наиболее крупная платформа 454 Life Sciences (в настоящее время приобретена Roche Diagnostics), может получать информацию о 400 миллионах нуклеотидах за 10-часовой период работы на одной установке. Каждый раунд работы стоит порядка пяти-семи тысяч долларов, что эквивалентно стоимости секвенирования генома млекопитающего в миллион долларов.
Литература
- Ronaghi M. (January 2001). «Pyrosequencing sheds light on DNA sequencing» (PDF). Genome Research 11: 3–11. DOI:10.1101/gr.11.1.3. PMID 11156611.
- Ronaghi et al. (2000). «Improved performance of Pyrosequencing using single-stranded DNA-binding protein». Analytical Biochemistry 286: 282. DOI:10.1006/abio.2000.4808. PMID 11067751.
- Elahi et al. (2004). «Pyrosequencing: a tool for DNA sequencing analysis». Methods Mol Biol 255: 211–219. PMID 15020827.
- Gharizadeh et al. (2002). «Long-read pyrosequencing using pure 2'-deoxyadenosine-5'-O'-(1-thiotriphosphate) Sp-isomer». Anal Biochem 301: 82. DOI:10.1006/abio.2001.5494. PMID 11811970.
- Fakhrai-Rad et al. (2002). «Pyrosequencing: an accurate detection platform for single nucleotide polymorphisms». Hum Mutat. 19: 479. DOI:10.1002/humu.10078. PMID 11968080.
Примечания
- ↑ Definition of pyrosequencing from the Nature Reviews Genetics Glossary. Архивировано из первоисточника 31 марта 2012. Проверено 28 октября 2008.
- ↑ Ronaghi et al. (1998-07-17). «A sequencing method based on real-time pyrophosphate». Science 281: 363. DOI:10.1126/science.281.5375.363. PMID 9705713.
- ↑ Ronaghi et al. (1996). «Real-time DNA sequencing using detection of pyrophosphate release». Analytical Biochemistry 242: 84. DOI:10.1006/abio.1996.0432. PMID 8923969.
- ↑ Nyrén, P. (2007). «The History of Pyrosequencing». Methods Mol Biology 373: 1–14. PMID 17185753.
- ↑ Qiagen acquires biosystems business from Biotage. Qiagen press release, October 1st, 2008
 Категории:
Категории:- Секвенирование
- Биотехнология
Wikimedia Foundation. 2010.